-
[Fastcluster] Fastcluster로 파이썬에서 seaborn clustermap 빨리 그리기Python/시각화 2023. 5. 8. 10:10반응형

Fastcluster를 이용해 파이썬에서 clustermap 빨리 그리기
파이썬(Pyhton)에서 속도 문제는 언제나 뜨거운 감자입니다. 못쓸 정도는 아니지만, 그렇다고 해서 쾌적한 속도를 보여주지도 않기 때문에 빅데이터 분석을 할 때는 언제나 속도 최적화 과정과 병렬 처리에 대한 지식을 잘 알아야 하기 때문입니다.
그리고, 이러한 노력은 다양한 프로그래머의 노력으로 더욱 편리한 패키지로 탄생하고 있습니다. 저는 최근에 클러스터맵(Clustermap)을 그릴 일이 생겼는데, 옛날 코드를 쓰려고 보니 특이한 안내메시지가 하나 보이더라고요. Fastcluster를 쓰면 Clustermap을 더 빨리 그릴 수 있다는 말이었습니다.
과연 얼마나 빨라질지 궁금해서, 제가 이전에 사용했던 클러스터맵 코드를 이용해 테스트를 해 봤습니다. 만약 이 코드를 따라하고 싶다면, 아래 코드 또는 깃허브에서 ipynb 파일을 다운로드한 뒤 실행해 보시기 바랍니다.
- Transcription profiling by high throughput sequencing from seedling, root, and floral buds of 17 Arabidopsis thaliana accessions 데이터
- Source: https://www.ebi.ac.uk/gxa/experiments/E-GEOD-53197/Downloads (TPM file)
# 패키지 불러오기 import pandas as pd import seaborn as sns from sklearn.ensemble import RandomForestClassifier import matplotlib.pyplot as pltara_17 = pd.read_csv('./Data/Arabidopsis_17/E-GEOD-53197-query-results.tpms.tsv', sep='\t', comment='#', index_col=1) ara_17 = ara_17.iloc[:, 1:] # 계산 편의상 missing value를 0으로 치환한다 ara_17 = ara_17.fillna(0) X = ara_17.values.T part = [x.split(', ')[1] for x in ara_17.columns] part = list(set(part)) # 중복되는 요소를 합친 후 리스트화 y = [part.index(x.split(', ')[1]) for x in ara_17.columns]rfc = RandomForestClassifier(n_estimators=2000, random_state=0) rfc.fit(X, y)RandomForestClassifier(n_estimators=2000, random_state=0)RandomForestClassifier(n_estimators=2000, random_state=0)m = rfc.feature_importances_ > 0 ara_17_fi = ara_17.index[m]sns.clustermap(ara_17.loc[ara_17_fi], standard_scale=0)
위 코드에서 마지막 셀을 실행하면 클러스터맵이 생성되는데, 여기에 걸리는 시간을 측정해 보겠습니다. jupyter에서는
%%timeit이라는 매직 커맨드(Magic Command)가 있는데, 이건%%timeit이 입력된 셀을 수 차례 실행한 뒤 소요시간의 평균을 측정해 주는 코드랍니다.참고로,
%%timeit은 특성상 코드를 여러 차례 실행하기 때문에, 생성된 클러스터맵이 잔뜩 쌓이지 않도록 클러스터맵이 그려진 뒤 지우도록plt.clf()함수를 맨 아래에 덧붙여 줬습니다. 어차피 도표를 그린 뒤 지우는 것이니 성능 테스트에 문제는 없답니다.%%timeit sns.clustermap(ara_17.loc[ara_17_fi], standard_scale=0) plt.clf()1.07 s ± 18 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)해당 클러스터맵을 그리는데는 1.07 s ± 18 ms 의 시간이 소요되었다고 합니다. 딱히 느리지는 않지만, 큰 데이터를 입력할 경우에는 기하급수적으로, 어쩌면 그 이상으로 느려질 수 있습니다. 그렇기 때문에, Fastcluster를 이용해서 한번 테스트를 해 보겠습니다. Fastcluster는 쉘 스크립트에
$ pip install fastcluster를 입력해 설치해 주시면 됩니다.%%timeit sns.clustermap(ara_17.loc[ara_17_fi], standard_scale=0) plt.clf()1.06 s ± 29.5 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)이번 클러스터맵 코드는 1.06 s ± 29.5 ms 의 시간이 소요되었다고 합니다. 사실상 속도의 차이를 보이지 않는것 같은데, 더욱 크기가 큰 데이터프레임을 입력한다면 유의미한 속도 차이가 날지도 모르겠습니다.
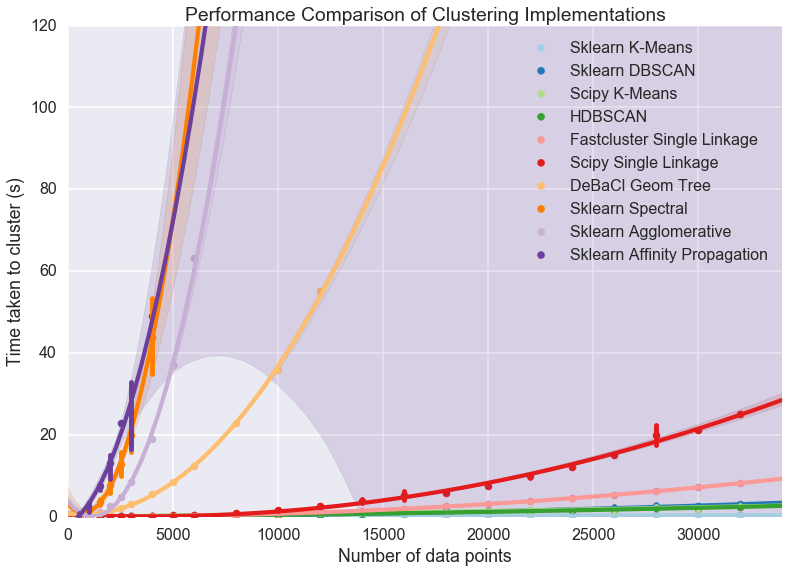
Fastcluster는 다른 벤치마크 결과를 살펴보면 규모가 큰 데이터에 대해서 상당히 빠른 속도를 보여줍니다. 저는 아마 다른 클러스터링 알고리즘과 별로 차이가 나지 않을 정도로 작은 데이터를 이용한 것으로 보이는데요, 자세한 내용은 아래 도표와 벤치마크 문서를 참고해 보시기 바랍니다.
 반응형
반응형'Python > 시각화' 카테고리의 다른 글
[Matplotlib] 파이썬 Matplotlib으로 이미지 읽고 그리기 (0) 2023.05.10 [Matplotlib] Matplotlib을 이용해 그래프 양식 설정하기 (0) 2023.04.28 [Matplotlib] Matplotlib을 이용해서 파이썬으로 그래프 그려보기 (0) 2023.04.27